こんにちは,米国データサイエンティストのかめ(@usdatascientist)です.
データサイエンスのためのPython入門第32回です(講座の目次はこちら).今回は大量のデータを処理する際に非常に重宝する”並列処理”について紹介します.
(「データサイエンスのためのPython講座」動画版がでました!詳細はこちら)
Pythonでは,multiprocessingというモジュールを使うことで超簡単に並列処理することができます.
時間がかかるループを使う場合は,並列処理が使えるか検討しましょう.(というより使える形に無理やりでもして,並列処理しましょう笑
目次
並列処理ってなに?
並列処理というのは,複数のプロセッサを使用して一つのタスクを細分化して並列に処理することです.
それぞれのプロセッサは並列に同時に処理するので,一つのプロセッサで処理した時に比べ当然早くなります.
プロセッサというのは処理を行う装置の総称ですが,Pythonで並列処理をする場合は(少なくとも本記事は)CPUを指します.
また,CPUには実際に処理をすることができるコアが複数入っています.(昔は1つしかありませんでしたが今は複数コアが主流.つまり厳密にいうと「コア」がここでの「プロセッサ」になります.)
なので複数のコアを使って並列処理していきます.
・・・そんなに難しい話じゃ無いですよね?笑 難しく聞こえますが,実際のコーディングはいたってシンプルですよ!
CPUとかコアなどの知識について勉強したい!という人は↓におすすめ本を書いているので読んでみてください.
並列処理の仕組みについて勉強しようとすると,結構奥が深いので大変です.データサイエンティストとして業務で並列処理を使うだけであれば,そこまでの知識は不要だと思います.
最初は本記事に書いてあるコードを毎回コピペして使える程度でOKです.そこから徐々に知識を広げるようにしましょう.
Pythonで並列処理をするには?→multiprocessingモジュールを使う
Pythonには簡単に並列処理を可能にする「multiprocessing」というモジュールがあります.もう本当に驚くほど簡単に並列処理ができちゃうんです..いい時代になりましたね←
本記事ではこのmultiprocessingモジュールでの並列処理のやり方について紹介していきます.このモジュールの難しいところは,同じことをやるのにいろんな書き方ができてしまうところです.なので初学者の人がこのモジュールについて調べると,いろんな人がいろんなやり方を紹介していて「?」になります.
ここでは私が最終的に行き着いた(落ち着いた)書き方を紹介していきます.
どれも私が実際の業務で普段使っている書き方です.参考にしていただければと思います.
- multiprocessingモジュールをインポートする
multiprocessingモジュールのPoolクラスをインポートして,Poolオブジェクトを使うことで並列処理を書いていきます.
なお,multiprocessingモジュールはPythonにすでに入っているモジュールなのでインストールは不要です.
|
1 |
from multiprocessing import Pool |
- Poolオブジェクトのインスタンスを作る
Poolクラスを使って,Poolオブジェクトのインスタンスを作ります.このインスタンスを使って,並列処理のコードを書いていきます.
インスタンス生成時に,processes引数に使用したいプロセッサの数を指定します.
|
1 2 |
cpu_num = 4 p = Pool(processes=cpu_num) |
cpu_numとしていますが,実際はコア数です.
- Macでコア数を確認する
自分のマシンがいくつのコアを持っているのか確認しておきましょう
コアには物理コアと論理コアという考え方があります.
単一のコアが複数のタスクを同時に処理する技術があるんですが(Hyper Threadingと言います),もしコアが一つしかなくても二つのタスクを同時に処理できるなら,利用者からみたら「2つのコアがある」のと同じことですよね?
この「物理的に存在するコア」のことを「物理コア」といい,Hyper Threadingを使った擬似的なコアのことを「論理コア」と言います.
現在でも多くのCPUがHypyer Threadingの技術を用しており,大抵のコンピュータでは物理コア数と論理コア数が異なります.
Macで物理コア数と論理コア数をそれぞれ見てみましょう.それぞれ以下のコマンドで確認できます.
|
1 2 3 4 |
$ sysctl -n hw.physicalcpu_max 6 $ sysctl -n hw.logicalcpu_max 12 |
$sysctl -n hw.physicalcpu_max で物理コアを, $sysctl -n hw.logicalcpu_max で論理コアの数を確認できます.
私のマシンでは物理コアが6個,論理コアが12個あります.
Poolクラスのprocesses引数には,物理コアの数を指定します.私の場合は最大6まで指定でき,7以上を指定するとうまく動作しなくなったり遅くなる場合があるようです.(stackoverflowの質問を参考)
multiprocessingモジュールの cpu_count() 関数を使うことでも物理コア数を取得することができます.
|
1 2 |
from multiprocessing import cpu_count cpu_count() |
|
1 |
6 |
この関数の返り値をPool()クラスのprocesses引数に指定することで,動的にプロセッサの数を指定できます.全てのCPUを占用するのは危険なので,以下のように指定することが多いです.
|
1 2 |
from multiprocessing import Pool, cpu_count p = Pool(processes=cpu_count() - 1) |
それでは,このpインスタンスを使って並列処理のコードを書いていきましょう!
p.map(fun, iterable)がもっとも基本的な使い方
最も基本的な使い方は,p.map()関数に処理を書いた関数とiterable(ほとんどのケースはリスト)を入れるやり方です.
map関数というのは並列処理以外にも使う一般的な関数で,Python特有のものでもなく,多くのプログラミング言語に標準装備されている関数です.
Pythonにも普通のmap関数があるので,まずは普通のmap関数の使い方を紹介します.
・map()関数の使い方
自分はRubyではよく使ってましたが,Pythonでは並列処理以外で使うことがほとんどないです.
map()関数は第一引数に関数,第二引数にイテラブル(iiterable)を入れます. iterableというのはイテレータ(iterator)になりうるオブジェクトです.リストやタプルはiterableオブジェクトです.(for文でイテレーションできますもんね)
初学者であればあまり用語を気にする必要はないです.データサイエンティストが業務でプログラミング系の用語を使うことは少ないです.(class, function, objectくらい使い分けれればOK) コードを書いていくうちに違いがわかってきます.
逆に統計や機械学習の用語はしょっちゅう会話で使うので注意です.
map()関数は,第二引数に指定したiterable(例えばリスト)のそれぞれの値にfunctionを適用した結果をリストで返します.
...意味わからないですよね?w コードを実行した結果を見れば一発で理解できると思います.
|
1 2 3 4 5 6 7 |
def double(n): return 2 * n params = [1, 2, 3, 4] results = map(double, params) #resultsはイテレータなのでlist()に入れて中身を表示 list(results) |
|
1 |
[2, 4, 6, 8] |
値を二倍にする double() 関数と,数字のリストをmap関数に入れると,それぞれの値が二倍になって返ってきています.
map() の返り値はイテレータなので,list()に入れて中身を表示しました.なんとなくわかっていただけたでしょうか?
multiprocessingモジュールを使うとmapでの処理を並列処理してくれます.データが長くなったり,重たい処理をする場合は基本的にはmultiprocessingを使うので,単体でmap()を使うケースというのはあまりないと思います.(for文を使う場合がほとんどだと思います.)
・p.map()関数で並列map処理
p.map()関数を使うことで通常のmapを並列に処理することが可能です.
使い方は一緒です.
並列処理をする場合,タスクを分割してそれぞれのCPUに投げてその結果を最後結合して...という処理をするので,それぞれのタスクが簡単だったりデータ数が短い場合は並列処理をしない方が早い事があります.
今回は,第30回の最後に紹介した「大量のpngデータをnpyデータに変換して保存する」処理を並列処理を使って実装したいと思います.かなり時間がかかる処理ですからね・・・!
(*データの準備は第29回 を参考にしてください.)
以下のコードが第30回で使ったコードです.やってみるとわかりますが,かなり時間がかかります.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
import cv2 import numpy as np from tqdm import tqdm # NumPy Arrayをsaveするフォルダ save_base_path = 'gaussian_filtered_nparrays/' for p in tqdm(glob('gaussian_filtered_images/*/*.png')): #pngデータをopencvでロード im = cv2.imread(p) #pngのファイル名をそのまま.npyファイルのファイル名にする file_name = p.split('/')[-1].split('.png')[0] save_path = save_base_path + file_name #全てのnpyファイルを同じフォルダに格納 np.save(save_path, im) |
(実行するにはgaussian_filtered_nparraysフォルダをカレントディレクトリに作成してください.)
このコードをp.map()を使って,以下のように書き換えることで並列処理にすることができます.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 |
import os import numpy as np import cv2 from multiprocessing import Pool, cpu_count from tqdm import tqdm from glob import glob p = Pool(processes=cpu_count()-1) # NumPy Arrayをsaveするフォルダ save_base_path = 'gaussian_filtered_nparrays_multi/' # フォルダ作成 if not os.path.exists(save_base_path): os.makedirs(save_base_path) def save_npy(path): #pngデータをopencvでロード im = cv2.imread(path) #pngのファイル名をそのまま.npyファイルのファイル名にする file_name = path.split('/')[-1].split('.png')[0] save_path = save_base_path + file_name #全てのnpyファイルを同じフォルダに格納 np.save(save_path, im) path_list = glob('gaussian_filtered_images/*/*.png') p.map(save_npy, path_list) # 並列処理が終わったら閉じます p.close() p.join() |
少し長いコードですが,今回新しく出てきた箇所は p.map(save_npy, path_list)の部分です.save_npyという関数をあらかじめ作っておき,glob()で取得したパスのリストをp.map()で並列処理していきます.
並列処理をしないやり方(第30回)に比べたら断然早いと思います.
p.close()で「もうpインスタンスにデータは渡さないよ」と明示し,並列処理を終了させます.実際にプロセスを終了させるにはp.join()を呼びます.Jupyterでコードを書くといつまでもメモリにpインスタンスが残るので,このようにp.close()->p.join()をコールすることで処理を正しく閉じることをお勧めします.(別ファイルでスクリプトを書く際は不要です.)
p.map()ではなくp.imap()を使う
p.map()ではなくp.imap()を使うことも多いです.私はこちらの方をよく使います.というかほぼimap()のほうが有利なケースだと思うのでこちらを使ってください(笑)
map()とimap()の違いを一言で言うと,imapは他の処理が終わってなくても次の処理を進める事ができます.いわゆるnon-blocking処理というやつです.そのため速度も早くメモリ効率も上がります.
先ほどのコードのp.map()の部分を以下のように書き換えてください.今回はtqdmも合わせて使います.tqdmについては第30回を参考にしてください.
|
1 |
list(tqdm(p.imap(save_npy, path_list), total=len(path_list))) |
p.imapの返り値はイテレータなので,total引数を渡す事でデータの総数をtqdmに教えてあげましょう.
tqdmもイテレータを返しますが,list()に入れる事でイテレーションが終わるのを待ち,結果を表示します.(この場合プログレスバー)
・・・あまり深く考えず,この型を覚えるのがいいと思います.
複数の引数を関数に渡す場合
先ほどまでの例だと,引数に入るのがimap()関数の第二引数に渡しているリストの値のみでした.
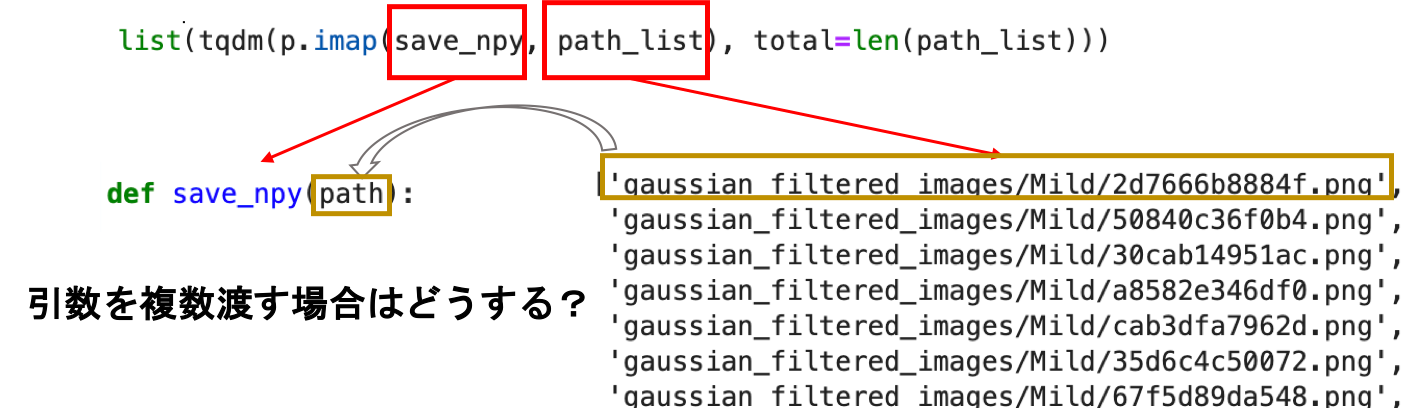
複数の引数を渡す関数にする場合,以下のようにラッパー関数を作ります.ラッパーというのはwrapする関数,つまり他の関数を包んで別の関数のように扱うものです.
今回はファイル名のsuffixにtype名を付け加えて保存してみます.(‘Mild’とか’Moderate’)
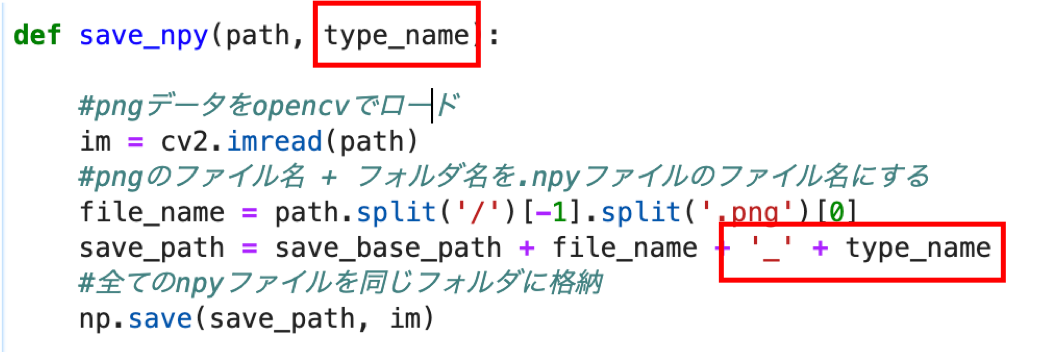
まず,save_npyを以下のように書き換えます.新しくtype_name引数をとり,ファイル名に追記します.
|
1 2 3 4 5 6 7 8 9 |
def save_npy(path, type_name): #pngデータをopencvでロード im = cv2.imread(path) #pngのファイル名 + フォルダ名を.npyファイルのファイル名にする file_name = path.split('/')[-1].split('.png')[0] save_path = save_base_path + file_name + '_' + type_name #全てのnpyファイルを同じフォルダに格納 np.save(save_path, im) |
下の赤の部分を追加しました!
さて,引数として渡すデータを準備します.通常タプルのリストを渡します.
今回は第一引数がファイルパス(path), 第二引数がフォルダ名(type_name)なので(path, type_name)のタプルをリストにしたものを作ります.
フォルダ名(type_name)を取得するには path.split('/')[1] でOKでしたね.(第29回でも同じことをしています.)これを使って以下のようにリストを使っています.
|
1 2 3 |
path_list = glob('gaussian_filtered_images/*/*.png') job_args = [(path, path.split('/')[1]) for path in path_list] job_args |
リストの内包表記を使っています.この辺あやしい人は第4回を読んでください!
このまま p.imap(save_npy, job_args)を実行してもダメです.なぜなら,job_argsの中にはあくまでもタプルのリストが入っていて,save_npy関数に渡されるのは一つのタプル(path, type_name)であってpathとtype_nameが別々に渡される訳ではありません.
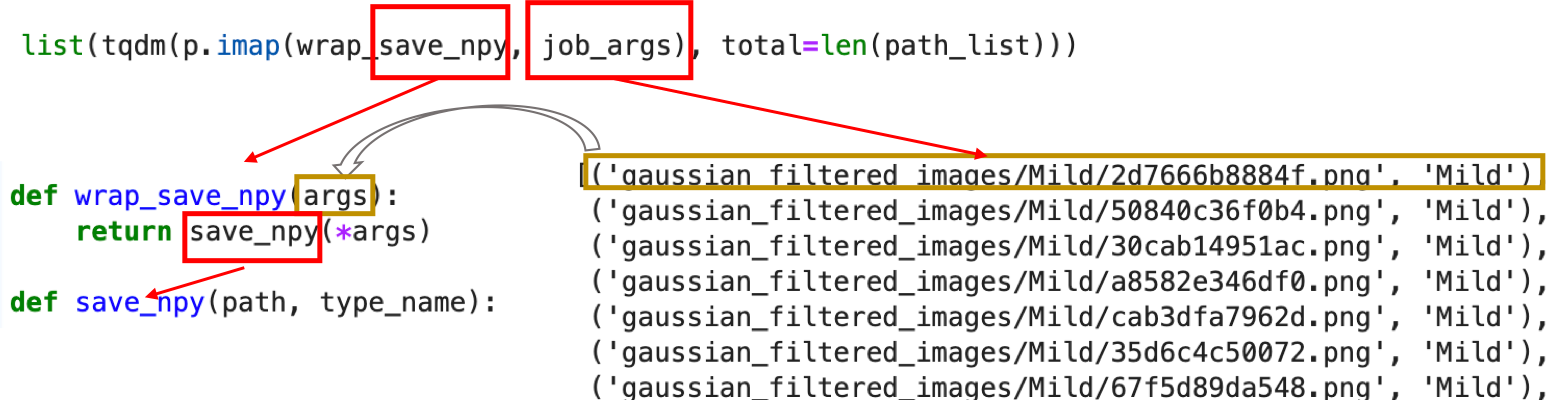
一方save_npy関数は先ほど引数を2つ受け取るようにしています.ここで,save_npy関数をラッピングするwrap_save_npy関数を作ります!
|
1 2 |
def wrap_save_npy(args): return save_npy(*args) |
*argsをうまく使って,1つ引数を複数の引数としてsave_npy関数に受け渡しています.(*argsについては第5回で説明しています.)
さーーーーーて
長くなってきましたねw
これで list(tqdm(p.imap(wrap_save_npy, job_args), total=len(path_list))) を呼べば完成です.
お疲れ様でした.
全体のコードは以下のようになります.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 |
import os import numpy as np import cv2 from multiprocessing import Pool, cpu_count from tqdm import tqdm from glob import glob p = Pool(processes=cpu_count()-1) # NumPy Arrayをsaveするフォルダ save_base_path = 'gaussian_filtered_nparrays_multi_w_type/' # フォルダ作成 if not os.path.exists(save_base_path): os.makedirs(save_base_path) def wrap_save_npy(args): return save_npy(*args) def save_npy(path, type_name): #pngデータをopencvでロード im = cv2.imread(path) #pngのファイル名 + フォルダ名を.npyファイルのファイル名にする file_name = path.split('/')[-1].split('.png')[0] save_path = save_base_path + file_name + '_' + type_name #全てのnpyファイルを同じフォルダに格納 np.save(save_path, im) path_list = glob('gaussian_filtered_images/*/*.png') job_args = [(path, path.split('/')[1]) for path in path_list] list(tqdm(p.imap(wrap_save_npy, job_args), total=len(path_list))) p.close() p.join() |
今回はフォルダ名を変えて,’gaussian_filtered_nparrays_multi_w_type’としました.
ここまでくると本当にいろんな知識を使っています.OpenCVやglob, split()などの文字列操作, osモジュールでのフォルダ作成,multiprocessingと盛りだくさんです.
少し難しく感じるかもしれませんが,Pythonを使うデータサイエンティストは日頃こういうコードを書いています.
私がPythonで並列処理をする際のコードは9割はこの形です.この形を覚えておけば,ほとんどのケースで対応できると思います.
コードを実行して,ファイル名 ‘pngファイル名_フォルダ名.npy’になっていることを確認しましょう!
ちなみに今回のコードを簡単に図に書くと以下のようになります.
まとめ
やはり一回の記事で並列処理をまとめるのは大変でしたw 思った以上に長くなってしまいましたが,本記事の内容だけでほとんどのケースを乗り切れると思います.
とにかく言いたいのは
最初はとにかくコピペでOK!!!!!
ということ.最初から理解してこのコードをかける必要はないです.
私も最初ずっとコピペしてましたよ.何度か使ってるうちに「もう少し理解しよう」という気持ちになると思います.
製品開発に組み込むコードでなければ,コピペで動けば問題ないです.
そんな気持ちでやってください.
今回の内容をまとめると
- from multiprocessing import Pool, cpu_count が基本のインポート
- p = Pool(processes=cpu_count() - 1) でPoolオブジェクトのインスタンスを作成 processes引数には使用する物理コアの数を入れる. cpu_count()-1 とすれば現在使用可能の物理コア-1個を入れることができる
- p.imap(func, iterable) で並列処理が可能
- p.join() と p.close() により並列処理を閉じる
- list(tqdm(p.imap(func, iterable))) とすることでプログレスバーを表示可能
- 複数の引数を渡す必要がある場合はラッパー関数を作成
この辺りのレベルになると,USで働くデータサイエンティストでも書けない/知らない人がちらほらいるレベルです.日本語の情報も少ないと思います.
しかし,実際の業務でめちゃくちゃ使えます.特に大量のファイルを処理する際は必須だと思いますし,並列処理は仕事の効率に直結するので知っておくといいと思います.
次回はついに最終回です!それでは!
追記)次回(最終回)書きました.今までJupyterに全部関数を定義してましたが,それだと今後行き詰まります.Pythonファイルを作って,そこに関数を定義して,Jupyterで使う方法を紹介します.基本的にはこのやり方でコーディングすることをおすすめします.